COVID-19 Prov-PER
Analisis de los datos reportados por el MINSA sobre la epidemioa de COVID-19 en Peru por provincia.
Datos Handbook Covid-19 Perú
Informacion Adicional Gobierno del Peru
Situacion Nivel Mundial Coronavirus COVID-19 Global Cases by the Center for Systems Science and Engineering (CSSE)
Actualizado: Abril 11, 2020
Loading packages
rm(list=ls())
library(tidyverse)
library(rio)
library(lubridate)
library(zoo)
library(readxl)
#library(ggthemr)
#ggthemr('dust')
library(colorspace)
#devtools::install_github("Financial-Times/ftplottools")
#library(ftplottools)
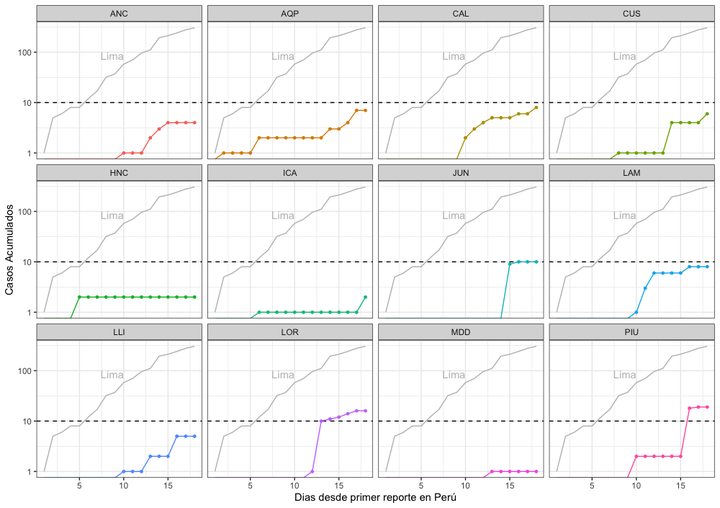
library(ggrepel)[Grafico 1: PCR]
rm(list=ls())
source("./_helpers/covid_prov-per_helpers.R")
dat <- read_excel("./_dat/reportes_minsa.xlsx", sheet=3) %>%
mutate(PositivosImputados_PCR = ifelse(is.na(PositivosImputados_PCR_PR),PositivosImputados_PCR,
PositivosImputados_PCR + PositivosImputados_PCR_PR)) %>%
dat_proc(init = 0)
dep <- dat[[2]]
trend <- dat[[3]]
a <- dat[[4]]
f1<-dep %>%
ggplot(aes(Día, acumulado, col=REGION)) +
#geom_hline(aes(yintercept = 100), linetype = "dashed") +
geom_line() +
geom_point(data = . %>% dplyr::filter(acumulado>0), size=1) +
geom_line(data = trend, col="gray") +
scale_y_log10() +
ylab("Casos Acumulados (PCR)") +
xlab(expression("Días desde primer reporte de casos en cada Región" %->% "")) + # *** CAMBIAR TITULO (init)
guides(col = F) +
#scale_color_discrete_sequential(palette = "Viridis") +
geom_text(data = . %>% group_by(REGION) %>% slice(n()),
aes(label = acumulado), nudge_y = -0.2, hjust=-.2) +
geom_text_repel(data = a,
aes(label = paste0("Lima: " , acumulado)), col = "gray", nudge_y = 0.3, hjust=1.5) +
geom_point(data = a, col= "gray") +
ggtitle(paste0("Casos (PCR) por Region. Actualizado hasta: ", Sys.Date())) +
facet_wrap(.~REGION, ncol = 4) +
theme_bw(base_size = 10)
#ft_theme(base_size = 10)
f1
[Grafico 2: totales = CR + Serologicas]
rm(list=ls())
source("./_helpers/covid_prov-per_helpers.R")
dat <- read_excel("./_dat/reportes_minsa.xlsx", sheet=3) %>%
dat_proc(init = 0, var = PositivosImputados_totales)
dep <- dat[[2]]
trend <- dat[[3]]
a <- dat[[4]]
f2<-dep %>%
ggplot(aes(Día, acumulado, col=REGION)) +
#geom_hline(aes(yintercept = 100), linetype = "dashed") +
geom_line() +
geom_point(data = . %>% dplyr::filter(acumulado>0), size=1) +
geom_line(data = trend, col="gray") +
scale_y_log10() +
ylab("Casos Acumulados (PCR + PR)") +
xlab(expression("Días desde primer reporte de casos en cada Región" %->% "")) + # *** CAMBIAR TITULO (init)
guides(col = F) +
#scale_color_discrete_sequential(palette = "Viridis") +
geom_text(data = . %>% group_by(REGION) %>% slice(n()),
aes(label = acumulado), nudge_y = -0.2, hjust=-.2) +
geom_text_repel(data = a,
aes(label = paste0("Lima: " , acumulado)), col = "gray", nudge_y = 0.3, hjust=1.5) +
geom_point(data = a, col= "gray") +
ggtitle(paste0("Casos Totales (PCR + PR) por Region. Actualizado hasta: ", Sys.Date())) +
facet_wrap(.~REGION, ncol = 4) +
theme_bw(base_size = 10)
#ft_theme(base_size = 10)
f2
[Grafico 3: fallecidos]
rm(list=ls())
source("./_helpers/covid_prov-per_helpers.R")
m <- read_excel("./_dat/_old/reportes_minsa_repo.xlsx", sheet = 4) %>%
group_by(Departamento, `Fecha de Reporte`) %>%
count() %>%
mutate(Fecha = as.Date(`Fecha de Reporte`)) %>%
ungroup() %>%
complete(Fecha = seq.Date(min(Fecha), max(Fecha), by="day"), nesting(Departamento),
fill = list(n = 0)) %>%
group_by(Departamento) %>%
mutate(Fallecidos_prev = cumsum(n),
Fecha = as.POSIXct(Fecha)) %>%
rename(REGION = Departamento) %>%
dplyr::select(-n, -`Fecha de Reporte`)
dat <- read_excel("./_dat/reportes_minsa.xlsx", sheet=3) %>%
full_join(m, by = c("REGION", "Fecha")) %>%
mutate(Fallecidos = ifelse(is.na(Fallecidos), Fallecidos_prev, Fallecidos)) %>%
dat_proc(init = 0, var = Fallecidos)
dep <- dat[[2]]
trend <- dat[[3]]
a <- dat[[4]]
f3<-dep %>%
ggplot(aes(Día, acumulado, col=REGION)) +
#geom_hline(aes(yintercept = 100), linetype = "dashed") +
geom_line() +
geom_point(data = . %>% dplyr::filter(acumulado>0), size=1) +
geom_line(data = trend, col="gray") +
scale_y_log10() +
ylab("Fallecidos Acumulados") +
xlab(expression("Días desde primer reporte de fallecidos en cada Región" %->% "")) + # *** CAMBIAR TITULO (init)
guides(col = F) +
#scale_color_discrete_sequential(palette = "Viridis") +
geom_text(data = . %>% group_by(REGION) %>% slice(n()),
aes(label = acumulado), nudge_y = 0.2, hjust=1) +
geom_text_repel(data = a,
aes(label = paste0("Lima: " , acumulado)), col = "gray", nudge_y = 0.3, hjust=1.5) +
geom_point(data = a, col= "gray") +
ggtitle(paste0("Fallecidos por Region. Actualizado hasta: ", Sys.Date())) +
facet_wrap(.~REGION, ncol = 4) +
theme_bw(base_size = 10)
#ft_theme(base_size = 10)
f3